Aulas Teórico-Práticas de Bioquímica
|
| Bases de Dados |
| Alinhamento de sequências |
| OMIM |
| Visualização de proteínas 2 |
| Árvores Filogenéticas |
Bases de Dados de Sequências de Nucleótidos
Este módulo vai apresentar, através de exemplos, algumas das Bases de dados de sequências de nucleótidos de acesso livre na internet.
As Bases de dados de sequências de nucleótidos são grandes repositórios onde são armazenados grandes volumes de informação aceitando sequências de nucleótidos da comunidade científica.
Apesar do grande avanço que representa toda esta informação disponível livremente, a tarefa de analisar e retirar informação destas Bases de dados pode ser difícil devido não só à grande quantidade de informação mas também ao facto de esta informação não estar apresentada de uma forma muito uniforme. De facto as sequências podem ser obtidas de diferentes fontes (cDNA ou DNA genómico), apresentar diferentes qualidades (versão inicial ou final) ou então são obtidas a partir de versões completas ou incompletas de um determinado genoma.
Outra dificuldade quando se trabalha com estas Bases de dados são os diferentes formatos em que as sequências são apresentadas. Para além disso, como existem diferentes Bases de dados para a o mesmo tipo de informação surge muitas vezes a necessidade de procurar uma determinada sequência em vários locais.
Para amenizar estas dificuldades existe uma colaboração entre as Bases de dados de acesso livre mais importantes para que a informação seja constantemente actualizada e sincronizada entre elas. Deste esforço surgiu assim o "Internacional Nucleotide Sequence Database Collaboration" que congrega as três Bases de dados mais importantes:
América: Entrez Nucleotides database (GenBank), pertence à pubmed: http://www.ncbi.nlm.nih.gov/entrez/query.fcgi?db=Nucleotide&itool=toolbar
Europa: EMBL Nucleótide Sequence Database
http://www.ebi.ac.uk
Ásia: DDBJ (DNA Data Bank of Japan)
http://www.ddbj.nig.ac.jp/
O tutorial que segue vai utilizar a Entrez como Base de dados exemplo. Mas é importante salientar que as 3 Bases de dados apresentadas acima estão sincronizadas excepto para sequências que foram adicionadas nas últimas 24 horas.
Módulo
1. Abrir um programa de navegação na Internet como o "Microsoft Internet Explorer" ou o "Mozilla Firefox" (utilizado neste tutorial). Abrir o site da NCBI (National Center for Biotechonology Information), http://www.ncbi.nlm.nih.gov/Entrez/.

O site da NCBI é um dos mais utilizados em trabalhos na área da Bioinformática. Apresenta muitos recursos de livre acesso nomeadamente várias Bases de dados bem como ferramentas para pesquisar e tratar os resultados obtidos nas nossas pesquisas.
2. Clicar em "All Databases" na barra azul indicada pela seta vermelha.
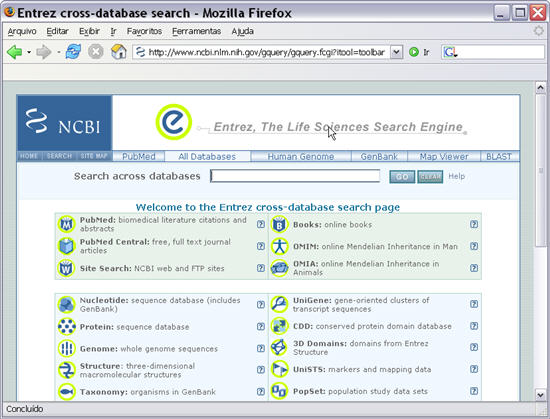
Ao executar o passo 2 deste tutorial surge o site "Entrez". Este site é uma tentativa da NCBI de integrar as Bases de dados mais importantes num só página. Na prática uma procura neste site origina os mesmos resultados do que se executarmos uma pesquisa no site inicial do NCBI, mas no "Entrez" poderá fazer uma pesquisa em várias Bases de dados ao mesmo tempo.
3. Digitar "Down Syndrome [Ti]"no campo de procura que se encontra à direita de "Search across databases", como indicado pela seta azul. Primir "Go".
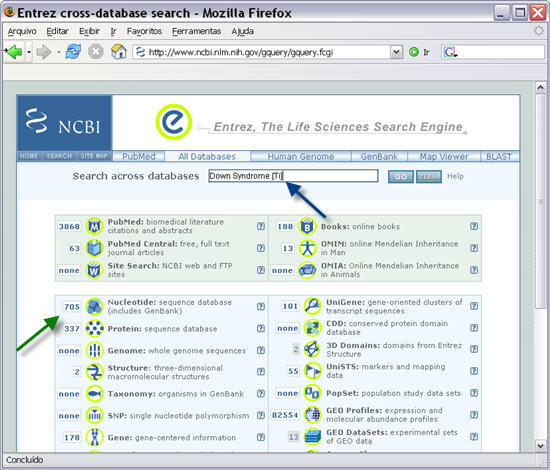
Como se pode ver na figura, a pesquisa é realizada em várias Bases de dados e o número de resultados é apresentado à esquerda de cada recurso pesquisado. Por exemplo na Base de dados de sequências de nucleótidos "Nucleotide" obtivemos 551 resultados. Na imagem temos 705 resultados (seta verde) porque representa uma pesquisa no ano passado e entretanto a apresentação de resultados foi modificada. A base de dados "Nucleotide" foi subdividida em duas:"CoreNucleotide" e "ESTs". Já a base de dados "Protein", de sequências de proteínas, apresenta 477 resultados contra 337 do ano passado.
4. Primir na Base de dados de sequências de nucleótidos "Nucleotide".
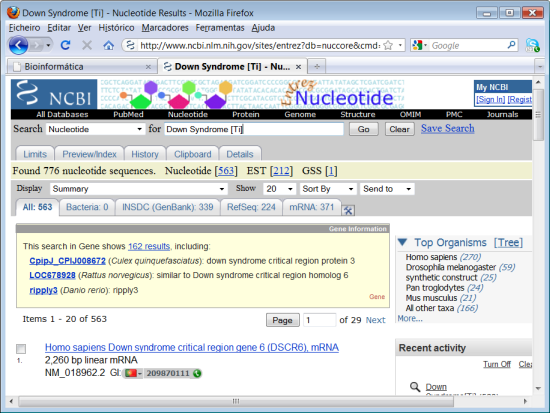
Nesta página podemos observar as sequências de nucleótidos que já foram de alguma forma relacionadas com o Syndrome de Down. No momento de execução deste tutorial as sequências são no total "All" 563; das quais 0 em bactérias "bacteria", 371 são RNAm ("mRNA") e 224 "RefSeq".
Quando se inicia uma procura deste género o problema principal é muitas vezes o número de resultados. Para diminuir este número vamos restringir a pesquisa.
5. Digitar "Down Syndrome [Ti] Homo Sapiens"no campo de procura. Primir "Go".
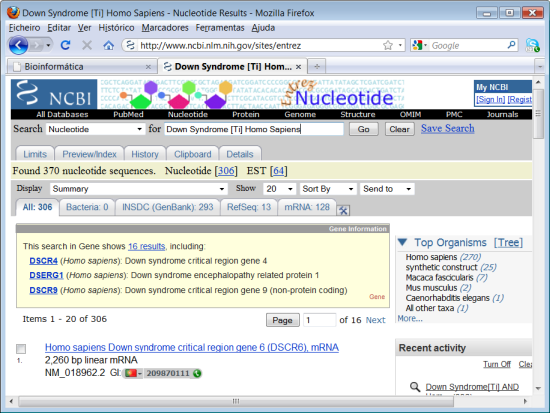
Como podemos observar os resultados indicados agora dizem respeito a sequências de nucleótidos de Homo Sapiens ao passo que antes apareciam resultados de Mus musculus (rato) entre outros organismos. Agora temos apenas 306 resultados.
6. Clicar em "This search in Gene shows 16 results, including".
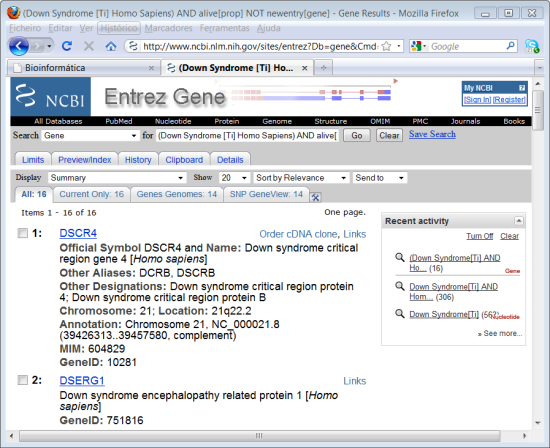
Nesta página podemos inspecionar os genes estudados que estão envolvidos de alguma forma no Sindrome de Down. Repare que mudou de Base de Dados, agora está na Base de Dados Gene onde podemos pesquisar de acordo com os genes conhecidos. Neste caso e como esperado temos vários genes envolvidos no Sindrome de Down pois estamos a falar de uma trissomia. Observe também que, como esperado, todos os genes se encontram no cromossoma 21.
7. Escolher o gene PIGP.
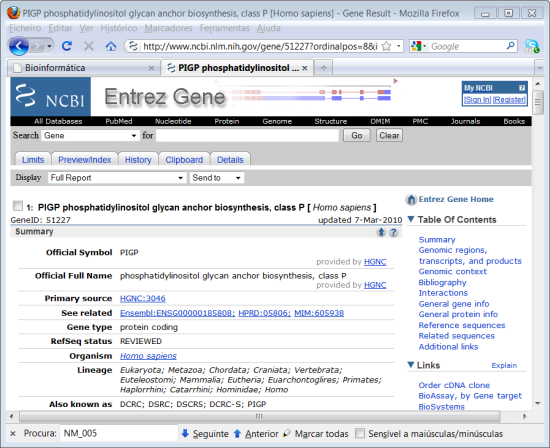
Nesta página temos um conjunto de informação muito variada sobre este gene: função, sinónimos (importante porque um gene pode muitas vezes ter muitos nomes diferentes) bibliografia e vários links para outras bases de dados. Ao ler o sumário podemos ter uma noção de qual o efeito potencial de existir uma cópia a mais deste gene em casos de Síndrome de Down.
8. Procurar o link com o nome NM 153682.2 que corresponde ao gene que estamos a observar. Ao clicar na sequência escolher opção GENBANK. Deverão observar a seguinte página.
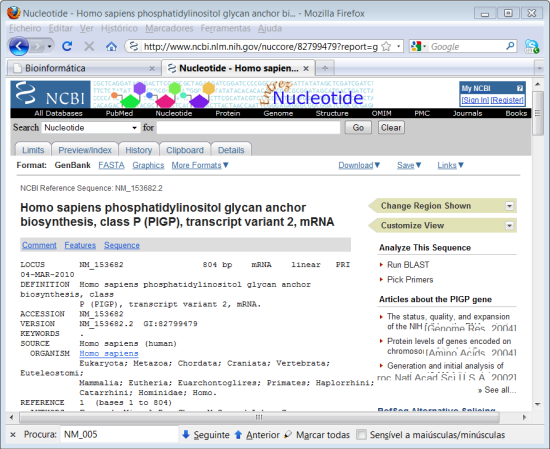
Estamos agora no base de dados "Nucleotide" uma vez que selecionamos uma sequência de DNA, neste caso o gene. A expressão NM_153682.2 corresponde ao código da sequência do gene e nesta página podes obter informação adicional detalhada sobre a sequência. Nesta caso diz-se que a sequência está anotada ("Anotation"). Para esta sequência também podemos escolher links relacionados com a sequência. Podemos consultar o artigo onde esta sequência foi originalmente publicada. Podemos também escolher outra Base de Dados, por exemplo a "OMIM", onde podemos verificar se esta sequência foi referenciada como estando relacionada com alguma doença.
Quantos pares de bases tem esta sequência?
E quantos aminoácidos tem a proteína após tradução deste gene?
Este gene tem mais transcriptos para além do apresentado?
9. No top da página onde está seleccionado "GenBank", escolher "FASTA".
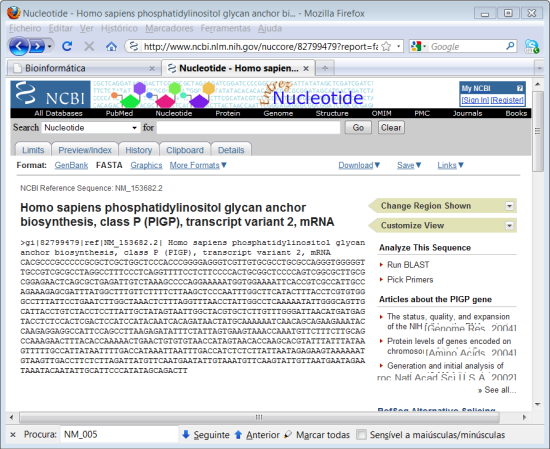
Com esta opção a página de internet com a nossa sequência pode agora ser gravada em formato texto (txt) e com a informação neste ficheiro em formato FASTA.
12. Clicar em "Download" e escolher opção "FASTA", escolher "Abrir com" e indicar que quer abrir o ficheiro como o WordPad.
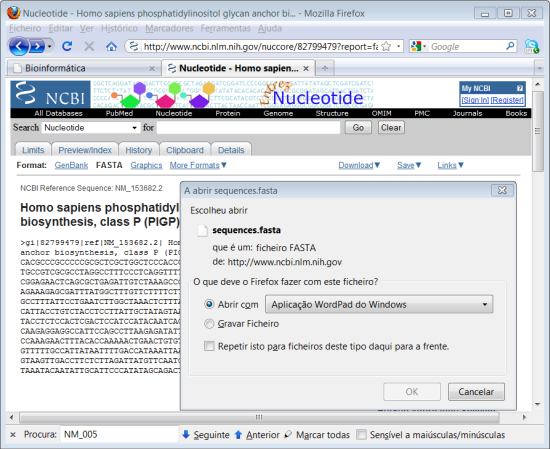
13. Clicar no menu "Ficheiro>Guardar como...". Escolher uma pasta para guardar o ficheiro e escolher um nome para o ficheiro. No exemplo demos o nome de "RCAN_sequence.txt". Clicar em Guardar.
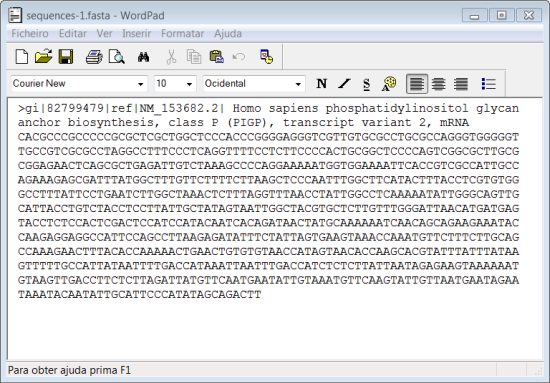
Não é boa ideia utilizar o Microsoft Word pois este software normalmente adiciona formatações. Estas formatações podem depois alterar a forma como outros programas lêm a sequência de nucleótidos.Se abrir com o "bloco de notas", a sequência guardada no formato FASTA é apresentada numa forma contínua o que por vezes torna impossível a visualização mais correcta da sequência. O WordPad é o software mais adequado para guardar sequências e normalmente existe em todos os computadores.
Agora vai utilizar esta sequência no módulo seguinte "Alinhamento de sequências"